Da quando questo articolo è stato scritto, abbiamo iniziato a esaminare le operazioni in tutto il mondo del Contractor USA nucleare/bio/chem , il preferito di Kusner-Trump, Battelle, e dei loro laboratori segreti in tutto il mondo.Da quando abbiamo dato il via, hanno iniziato a minacciare i nostri reporter. E questo è stato un grave errore.Inviate questo documento a qualsiasi medico o altro specialista qualificato in scienze biologiche. E vedete cosa dicono.

introduzione
I documenti di seguito mostreranno che la ricerca per creare COVID 19 è iniziata negli Stati Uniti nel 2006 ed è culminata in una bio-arma di successo nel 2015, con lavoro svolto presso l’Università della Carolina del Nord e ad Harvard e presso il laboratorio della Food and Drug Administration in Arkansas .
Il loro lavoro era intitolato:
Un gruppo simile alla SARS di coronavirus di pipistrello circolante mostra il potenziale di emergenza umana
Hanno fatto questo e altro, molto di più come leggerai di seguito.
Come diceva Trump, ancora e ancora, i cinesi erano coinvolti.
Laboratorio chiave di agenti patogeni speciali e biosicurezza, Istituto di virologia di Wuhan, Accademia cinese delle scienze, Wuhan, Cina ha fornito il virus del pipistrello di Wuhan, utilizzato nello studio americano. Il loro nome è stato incluso solo per quel motivo.
COVID 19 era un progetto di bio-armi dell’esercito americano per la produzione di una malattia che causava la polmonite che sarebbe quasi impossibile vaccinare nei pazienti di età superiore ai 40 anni.
La prova è qui, basta scorrere verso il basso. Lo studio è stato condotto dall’Università della Carolina del Nord e finanziato da USAID / CIA. Ha scelto un virus pipistrello cinese e ha scelto di includere anche una struttura medica a Wuhan.
Ora sappiamo perché, una cortina fumogena della colpa di un programma che la Cina aveva poco o nulla a che fare, qualcosa di satanicamente malvagio e puramente americano.
Nel novembre 2015 è stato pubblicato uno studio che delineava la capacità di produrre il virus con cui abbiamo a che fare ora. Tra i molti coinvolti c’era un laboratorio a Wuhan, in Cina. È stato elencato dall’inizio come una delle dozzine, principalmente americane, che lavorano a questo progetto.
Tuttavia, un partecipante chiave è stato escluso, USAID. Si sospetta, profondamente, che USAID sia un fronte per la ricerca americana sulla bio-guerra come quella condotta a Tbilisi, in Georgia e altrove, molto documentata. Questa è la citazione che aggiunge USAID al gruppo di finanziamento della ricerca.

Cambiare la storia
20 novembre 2015
Nella versione di questo articolo inizialmente pubblicata online, gli autori hanno omesso di riconoscere una fonte di finanziamento, il finanziamento USAID-EPT-PREDICT da EcoHealth Alliance, a Z.-LS. L’errore è stato corretto per le versioni stampate, PDF e HTML di questo articolo .
- Presenteremo ora l’articolo da parte della Pravda, di seguito, e lo studio reale che dimostra la capacità di produrre COVID 19, dimostrando che non è un virus naturale una volta per tutte.
- Per quanto riguarda chi ha fatto cosa, questo non è il nostro lavoro, ma stiamo dimostrando, categoricamente, che quando viene menzionato un laboratorio cinese, si tratta di un attore minore di uno sforzo americano, come indicato esaustivamente di seguito.
- Ciò rende la discussione del laboratorio di Wuhan probabilmente complice della guerra biologica.
- Allo stesso modo, quando la rivista Forbes e altri hanno dichiarato di poter provare che COVID 19 è stato creato in modo naturale , naturalmente, loro avevano lo stesso accesso che abbiamo noi, dunque sospettiamo che facciano parte di uno sforzo di disinformazione legato all’USAID e alla bio-guerra.
- Il sospetto non è una prova. La prova è provata ci sono abbastanza prove per annegarci dentro. I nostri ringraziamenti ai professionisti medici americani che si sono fatti valere dall’esercito americano e dalla CIA e che ci hanno aiutato a portarci dove siamo ora, una nazione spezzata.
Pravda.Ru: tale materiale è apparso nel 2015 sul sito web della rivista scientificaNatura nel 2015. Quindi gli autori hanno affermato che dopo l’avvento del virus SARS (2002-2003) e della Sindrome Respiratoria del Medio Oriente (MERS), gli scienziati erano a conoscenza del rischio di trasmissione interspecifica che porterebbe a un’epidemia tra le persone.
Esperimento di laboratorio riuscito
Tra le altre cose, il team di ricerca ha studiato i pipistrelli, che sono i maggiori incubatori di coronavirus. Tuttavia, i pipistrelli non potevano trasmettere il coronavirus all’uomo perché non potevano interagire con le cellule umane con i recettori ACE2.
Il materiale ha anche affermato che i pipistrelli a ferro di cavallo portano un ceppo di coronavirus SARS che può essere trasmesso all’uomo. È stato chiamato virus SHC014-CoV.
Per studiare meglio questo virus, gli scienziati hanno copiato il coronavirus e lo hanno infettato con cavie (topi) da laboratorio. I risultati hanno mostrato che il virus è davvero in grado di legarsi alle cellule umane con recettori ACE2 e moltiplicarsi nelle cellule del sistema respiratorio.
Nel lavoro di ricerca, si osserva che i materiali di laboratorio, i campioni e le attrezzature utilizzati nella ricerca sono stati ottenuti dall‘Istituto di Ricerca Medica delle Malattie Infettive dell’Esercito. Sebbene non sia ancora possibile affermare con certezza che il virus testato nei topi di laboratorio sia lo stesso del coronavirus SARS-Cove-2.
Politica della NATO
Tuttavia, cose interessanti possono essere trovate nei documenti precedenti.
Per esempio:
- Il rapporto di attività dell’Alleanza del 2019 afferma che nel 2019, il primo posto dell’Alleanza nella ricerca e nello sviluppo è stato occupato dal tema della radiochimica e della protezione biologica (29%), spostando il problema apparentemente più urgente dell’Europa – l’antiterrorismo (si è rivelato essere 4) – m priorità).
- Un anno prima, nel 2018, la situazione era esattamente l’opposto: il terrorismo, come dovrebbe essere, era al primo posto (28%) e la protezione radiochimica e biologica al quarto (13%).
Come scrive il boccino di Bruxelles nel canale dei telegrammi, “data l’assenza di ragioni visibili per un cambiamento così marcato negli interessi scientifici, ci sono due opzioni ed entrambe sono spiacevoli:
- o la NATO ora cancella il quinto punto, falsificando i dati per mostrare “e siamo sempre preparati ai virus, siamo moderni”,
- o anche nel 2019 nell’alleanza, Dio mi perdoni, sapevano da dove sarebbe venuto il problema.
Sì, la prima opzione è molto più reale, ma, vedi, i fatti sono sorprendenti.
Fonte: Pravda

Ricerca originale 2015 inedita e completa
Pubblicato: 09 novembre 2015Un gruppo simile alla SARS di coronavirus di pipistrello circolante mostra il potenziale di emergenza umana di Vineet D Menachery, Boyd L Yount Jr, Kari Debbink, Sudhakar Agnihothram, Lisa E Gralinski, Jessica A Plante, Rachel L Graham, Trevor Scobey, Xing-Yi Ge, Eric F Donaldson, Scott H Randell, Antonio Lanzavecchia, Wayne A Marasco, Zhengli-Li Shi, Ralph S Baric
Nature Medicine volume 21 , pagine 1508-1513 ( 2015 )
Una rettifica di questo articolo è stata pubblicata il 06 aprile 2016
Questo articolo è stato aggiornato
Abstract
L’emergenza della sindrome respiratoria acuta grave coronavirus (SARS-CoV) e della sindrome respiratoria del Medio Oriente (MERS) -CoV sottolinea la minaccia di eventi di trasmissione tra specie che portano a focolai nell’uomo. Qui esaminiamo il potenziale della malattia di un virus simile alla SARS, SHC014-CoV, che è attualmente in circolazione nelle popolazioni di pipistrelli a ferro di cavallo cinesi 1 . Utilizzando il sistema di genetica inversa SARS-CoV 2 , abbiamo generato e caratterizzato un virus chimerico che esprime il picco del coronavirus di pipistrello SHC014 in una spina dorsale SARS-CoV adattata dal mouse.
I risultati indicano che i virus del gruppo 2b che codificano il picco SHC014 in una spina dorsale di tipo selvatico possono utilizzare in modo efficiente più ortologi del recettore SARS nell’enzima II di conversione dell’angiotensina umana (ACE2), replicarsi efficacemente nelle cellule primarie delle vie aeree umane e ottenere titoli in vitro equivalenti a ceppi epidemici di SARS-CoV. Inoltre, esperimenti in vivo dimostrano la replicazione del virus chimerico nel polmone di topo con notevole patogenesi.
La valutazione delle modalità immunoterapiche e profilattiche basate sulla SARS ha rivelato scarsa efficacia; sia l’anticorpo monoclonale che l’approccio al vaccino non sono riusciti a neutralizzare e proteggere dall’infezione da CoV usando la nuova proteina spike. Sulla base di questi risultati, abbiamo sinteticamente ri-derivato un virus ricombinante SHC014 a lunghezza intera infettiva e dimostriamo una solida replicazione virale sia in vitro che in vivo . Il nostro lavoro suggerisce un potenziale rischio di riemersione di SARS-CoV da virus attualmente circolanti nelle popolazioni di pipistrelli.
Principale
L’emergere di SARS-CoV ha inaugurato una nuova era nella trasmissione tra le specie di gravi malattie respiratorie con la globalizzazione che porta a una rapida diffusione in tutto il mondo e un enorme impatto economico 3 , 4 . Da allora, diversi ceppi, compresi i ceppi di influenza A H5N1, H1N1 e H7N9 e MERS-CoV, sono emersi da popolazioni animali, causando notevoli malattie, mortalità e difficoltà economiche per le regioni colpite 5 . Sebbene le misure di salute pubblica siano state in grado di fermare l’epidemia di SARS-CoV 4 , recenti studi di metagenomica hanno identificato sequenze di virus simili a SARS strettamente correlati circolanti nelle popolazioni di pipistrelli cinesi che potrebbero rappresentare una minaccia futura 1 , 6 .
Tuttavia, i soli dati di sequenza forniscono informazioni minime per identificare e preparare futuri virus pre-pandemici. Pertanto, per esaminare il potenziale di emergenza (cioè il potenziale di infettare gli esseri umani) dei CoV di pipistrelli circolanti, abbiamo costruito un virus chimerico che codifica una nuova proteina spike di CoV zoonotica, dalla sequenza RsSHC014-CoV che è stata isolata dai pipistrelli a ferro di cavallo cinesi 1 — Nel contesto della spina dorsale adattata al mouse SARS-CoV. Il virus ibrido ci ha permesso di valutare la capacità della nuova proteina spike di causare la malattia indipendentemente da altre mutazioni adattative necessarie nella sua spina dorsale naturale.
Usando questo approccio, abbiamo caratterizzato l’infezione da CoV mediata dalla proteina di picco SHC014 nelle cellule primarie delle vie aeree umane e in vivo e testato l’efficacia delle terapie immunitarie disponibili contro SHC014-CoV. Insieme, la strategia traduce i dati metagenomici per aiutare a prevedere e prepararsi per i futuri virus emergenti.
Le sequenze di SHC014 e il relativo RsWIV1-CoV mostrano che questi CoV sono i parenti più vicini ai ceppi di SARS-CoV epidemici ( Fig. 1a, b ); tuttavia, ci sono differenze importanti nei 14 residui che legano l’ACE2 umano, il recettore per SARS-CoV, inclusi i cinque che sono fondamentali per l’intervallo ospite: Y442, L472, N479, T487 e Y491 (rif. 7 ).
Nel WIV1, tre di questi residui differiscono dal ceppo di SARS-CoV Urbani epidemico, ma non si prevedeva che alterassero il legame con ACE2 ( Figura 1a, b supplementare e Tabella 1 supplementare ). Questo fatto è confermato da entrambi gli esperimenti di pseudotipizzazione che hanno misurato la capacità dei lentivirus che codificano le proteine del picco WIV1 di entrare nelle cellule che esprimono ACE2 umano ( Figura 1 complementare ) e dai test di replicazione in vitro del WIV1-CoV (rif. 1 ). Al contrario, 7 su 14 residui di interazione ACE2 in SHC014 sono diversi da quelli in SARS-CoV, inclusi tutti e cinque i residui critici per l’intervallo ospite ( Figura 1c supplementare e Tabella 1 supplementare ).
Questi cambiamenti, associati al fallimento dei lentivirus pseudotipati che esprimono il picco SHC014 per entrare nelle cellule ( Figura 1d supplementare ), hanno suggerito che il picco SHC014 non è in grado di legare l’ACE2 umano. Tuttavia, cambiamenti simili nei ceppi SARS-CoV correlati erano stati segnalati per consentire l’associazione ACE2 7 , 8 , suggerendo che erano necessari ulteriori test funzionali per la verifica.
Pertanto, abbiamo sintetizzato il picco SHC014 nel contesto della spina dorsale SARS-CoV adattata per la cavia, adattata alla cavia (di seguito riferiamo al CoV chimerico come SHC014-MA15) per massimizzare l’opportunità di patogenesi e studi sui vaccini nelle cavie ( Figura complementare . 2a ). Nonostante le previsioni di entrambi gli esperimenti di modellazione e pseudotipizzazione basati sulla struttura, SHC014-MA15 era praticabile e replicato a titoli elevati nelle cellule Vero ( Figura 2b supplementare ). Simile alla SARS, SHC014-MA15 richiedeva anche una molecola ACE2 funzionale per l’ingresso e poteva usare ortologi ACE2 umani, zibetto e pipistrello ( Figura 2c, d supplementare ).
Per testare la capacità del picco SHC014 di mediare l’infezione delle vie aeree umane, abbiamo esaminato la sensibilità della linea cellulare epiteliale umana delle vie respiratorie Calu-3 2B4 (rif. 9 ) alle infezioni e abbiamo trovato una robusta replica SHC014-MA15, paragonabile a quella di SARS-CoV Urbani ( Fig. 1c ). Per estendere questi risultati, le colture epiteliali primarie delle vie aeree umane (HAE) sono state infettate e hanno mostrato una replicazione robusta di entrambi i virus ( Fig. 1d ). Insieme, i dati confermano la capacità dei virus con il picco SHC014 di infettare le cellule delle vie aeree umane e sottolineano la potenziale minaccia di trasmissione tra specie di SHC014-CoV.
Figura 1: i virus simili alla SARS si replicano nelle cellule delle vie aeree umane e producono patogenesi in vivo .
( a ) Le sequenze del genoma a lunghezza intera di CoV rappresentative sono state allineate e mappate filogeneticamente come descritto nei Metodi online . La barra di scala rappresenta sostituzioni nucleotidiche, con solo il supporto bootstrap superiore al 70% che viene etichettato. L’albero mostra i CoV divisi in tre gruppi filogenetici distinti, definiti come α-CoV, β-CoV e γ-CoV. I cluster di sottogruppi classici sono contrassegnati come 2a, 2b, 2c e 2d per i β-CoV e come 1a e 1b per i α-CoV. ( b ) Le sequenze di aminoacidi dei domini S1 dei picchi di β-CoV rappresentativi del gruppo 2b, incluso SARS-CoV, sono state allineate e mappate filogeneticamente. La barra della scala rappresenta le sostituzioni di aminoacidi. ( c , d) Replicazione virale di SARS-CoV Urbani (nero) e SHC014-MA15 (verde) dopo infezione di cellule Calu-3 2B4 ( c ) o colture cellulari HAE con interfaccia aria-liquido primaria ben differenziate ( d ) in una molteplicità di infezione (MOI) di 0,01 per entrambi i tipi di cellule. I campioni sono stati raccolti in singoli punti temporali con repliche biologiche ( n = 3) per entrambi gli esperimenti di Calu-3 e HAE. ( e , f ) Perdita di peso ( n = 9 per SARS-CoV MA15; n = 16 per SHC014-MA15) ( e ) e replicazione virale nei polmoni ( n = 3 per SARS-CoV MA15; n = 4 per SHC014- MA15) ( f) di topi BALB / c di 10 settimane infettati da 1 × 10 4 pfu di SARS-CoV MA15 (nero) o SHC014-MA15 (verde) adattato per topo attraverso la via intranasale (in). ( g, h ) Sono mostrate immagini rappresentative delle sezioni polmonari colorate per l’antigene SARS-CoV N da topi infetti da SARS-CoV MA15 ( n = 3 topi) ( g ) o SHC014-MA15 ( n = 4 topi) ( h ). Per ogni grafico, il valore centrale rappresenta la media del gruppo e le barre di errore definiscono le barre della scala sem, 1 mm.
Per valutare il ruolo del picco SHC014 nel mediare l’infezione in vivo , abbiamo infettato topi BALB / c di 10 settimane con 10 4 unità formanti placca (pfu) di SARS-MA15 o SHC014-MA15 ( Fig. 1e – h ). Gli animali infetti da SARS-MA15 hanno subito una rapida perdita di peso e letalità per 4 giorni dopo l’infezione (dpi); al contrario, l’infezione da SHC014-MA15 ha prodotto una sostanziale perdita di peso (10%) ma nessuna letalità nelle cavie ( Fig. 1e ). L’esame della replicazione virale ha rivelato titoli virali quasi equivalenti dai polmoni delle cavie infettate da SARS-MA15 o SHC014-MA15 ( Fig. 1f ). Considerando che i polmoni dei topi con infezione da SARS-MA15 hanno mostrato una colorazione robusta sia nei bronchioli terminali che nel parenchima polmonare 2 dpi ( Fig. 1g), quelli delle cavie con infezione da SHC014-MA15 hanno mostrato una ridotta colorazione dell’antigene delle vie aeree ( Fig. 1h ); al contrario, non è stato osservato alcun deficit nella colorazione dell’antigene nel parenchima o nel punteggio istologico complessivo, suggerendo un’infezione differenziale del tessuto polmonare per SHC014-MA15 ( Tabella supplementare 2 ). Successivamente abbiamo analizzato l’infezione in animali più sensibili, anziani (di 12 mesi). Gli animali con infezione da SARS-MA15 hanno perso rapidamente peso e hanno ceduto all’infezione ( Figura 3a, b supplementare ). L’infezione da SHC014-MA15 ha causato una perdita di peso robusta e sostenuta, ma ha avuto una minima letalità. Le tendenze nei modelli di istologia e colorazione dell’antigene che abbiamo osservato nei topi giovani sono state conservate negli animali più anziani ( Tabella Supplementare 3). Abbiamo escluso la possibilità che SHC014-MA15 stia mediando l’infezione attraverso un recettore alternativo sulla base di esperimenti usando topi Ace2 – / – , che non hanno mostrato perdita di peso o colorazione dell’antigene dopo l’infezione SHC014-MA15 ( Figura complementare 4a, b e Supplementare Tabella 2 ). Insieme, i dati indicano che i virus con il picco SHC014 sono in grado di indurre la perdita di peso nei topi nel contesto di una spina dorsale virale CoV.
Data l’efficacia preclinica delle terapie con anticorpi monoclonali di Ebola, come ZMApp 10 , abbiamo successivamente cercato di determinare l’efficacia degli anticorpi monoclonali SARS-CoV contro l’infezione da SHC014-MA15. Quattro anticorpi monoclonali umani ampiamente neutralizzanti destinati alla proteina spike SARS-CoV erano stati precedentemente segnalati e sono probabili reagenti per l’immunoterapia 11 , 12 , 13 . Abbiamo esaminato l’effetto di questi anticorpi sulla replicazione virale (espressa come inibizione percentuale della replicazione virale) e abbiamo scoperto che mentre il SARS-CoV di tipo selvaggio Urbani era fortemente neutralizzato da tutti e quattro gli anticorpi a concentrazioni di anticorpi relativamente basse ( Fig. 2a-d), la neutralizzazione variava per SHC014-MA15. Fm6, un anticorpo generato dalla visualizzazione dei fagi e dai mutanti di fuga 11 , 12 , ha raggiunto solo i livelli di fondo di inibizione della replicazione SHC014-MA15 ( Fig. 2a ). Analogamente, anche gli anticorpi 230.15 e 227.14, derivati dalle cellule della memoria B dei pazienti con infezione da SARS-CoV 13 , non riuscivano a bloccare la replicazione SHC014-MA15 ( Fig. 2b, c). Per tutti e tre gli anticorpi, le differenze tra le sequenze di aminoacidi a picco SARS e SHC014 corrispondevano a cambiamenti di residui diretti o adiacenti rilevati nei mutanti di fuga SARS-CoV (fm6 N479R; 230.15 L443V; 227.14 K390Q / E), che probabilmente spiega l’assenza degli anticorpi ‘attività neutralizzante contro SHC014. Infine, l’anticorpo monoclonale 109,8 è stato in grado di ottenere una neutralizzazione del 50% di SHC014-MA15, ma solo ad alte concentrazioni (10 μg / ml) ( Fig. 2d ). Insieme, i risultati dimostrano che gli anticorpi ampiamente neutralizzanti contro la SARS-CoV possono avere solo un’efficacia marginale contro i ceppi emergenti di CoV simili alla SARS come SHC014.
Figura 2: gli anticorpi monoclonali SARS-CoV hanno efficacia marginale nei confronti di CoV simili a SARS.
( a – d ) Saggi di neutralizzazione che valutano l’efficacia (misurata come riduzione del numero di placche) di un pannello di anticorpi monoclonali, tutti originariamente generati contro l’epidemia di SARS-CoV, contro l’infezione di cellule Vero con SARS-CoV Urbani (nero) o SHC014-MA15 (verde). Gli anticorpi testati erano fm6 ( n = 3 per Urbani; n = 5 per SHC014-MA15) 11 , 12 ( a ), 230.15 ( n = 3 per Urbani; n = 2 per SHC014-MA15) ( b ), 227.15 ( n = 3 per Urbani; n = 5 per SHC014-MA15) ( c ) e 109,8 ( n = 3 per Urbani; n = 2 per SHC014-MA15) 13 ( d ). Ogni punto dati rappresenta la media del gruppo e le barre di errore definiscono il sem Nota che le barre di errore nelle celle Vero con infezione da SARS-CoV Urbani in b , c sono sovrapposte dai simboli e non sono visibili.
Per valutare l’efficacia dei vaccini esistenti contro l’infezione da SHC014-MA15, abbiamo vaccinato topi anziani con SARS-CoV intero intero (DIV) a doppia inattivazione. Precedenti lavori hanno dimostrato che DIV potrebbe neutralizzare e proteggere i giovani topi dalle sfide con un virus omologa 14 ; tuttavia, il vaccino non è riuscito a proteggere gli animali anziani in cui è stata osservata anche una patologia immunitaria aumentata, indicando la possibilità che gli animali vengano danneggiati a causa della vaccinazione 15 . Qui abbiamo scoperto che DIV non ha fornito protezione dalle sfide con SHC014-MA15 per quanto riguarda la perdita di peso o il titolo virale ( Figura 5a, b supplementare ). Coerente con un precedente rapporto con altri gruppi eterologhi 2b CoVs 15, anche il siero di topi anziani vaccinati con DIV non è riuscito a neutralizzare SHC014-MA15 ( Figura 5c supplementare ). In particolare, la vaccinazione con DIV ha prodotto una robusta patologia immunitaria ( Tabella supplementare 4 ) ed eosinofilia ( Figura 5d-f supplementare ). Insieme, questi risultati confermano che il vaccino DIV non sarebbe protettivo contro l’infezione da SHC014 e potrebbe eventualmente aumentare la malattia nel gruppo vaccinato invecchiato.
Contrariamente alla vaccinazione delle cavie con DIV, l’uso di SHC014-MA15 come vaccino vivo e attenuato ha mostrato una potenziale protezione crociata contro la sfida con SARS-CoV, ma i risultati hanno importanti avvertenze. Abbiamo infettato topi giovani con 10 4 pfu di SHC014-MA15 e li abbiamo osservati per 28 giorni. Abbiamo quindi sfidato i topi con SARS-MA15 al giorno 29 ( Figura 6a supplementare ). La precedente infezione dei topi con la dose elevata di SHC014-MA15 conferiva protezione contro la sfida con una dose letale di SARS-MA15, sebbene ci fosse solo una risposta minima di neutralizzazione SARS-CoV dagli antisieri suscitati 28 giorni dopo l’infezione da SHC014-MA15 ( Figura 6b supplementare, 1: 200). In assenza di un potenziamento dell’antigene secondario, 28 dpi rappresenta il picco atteso dei titoli anticorpali e implica che nel tempo ci sarà una protezione ridotta contro SARS-CoV 16 , 17 . Risultati simili che mostrano protezione contro la sfida con una dose letale di SARS-CoV sono stati osservati nei topi BALB / c anziani rispetto alla perdita di peso e alla replicazione virale ( Figura 6c, d supplementare ). Tuttavia, la dose di infezione SHC014-MA15 di 10 4 pfu ha indotto una perdita di peso> 10% e letalità in alcuni animali anziani ( Fig. 1 e Fig. 3 supplementare). Abbiamo scoperto che la vaccinazione con una dose più bassa di SHC014-MA15 (100 pfu), non ha indotto la perdita di peso, ma non è riuscita a proteggere gli animali anziani da una sfida di dose letale SARS-MA15 ( Figura 6e, f supplementare ). Insieme, i dati suggeriscono che la sfida SHC014-MA15 può conferire una protezione crociata contro SARS-CoV attraverso epitopi conservati, ma la dose richiesta induce la patogenesi e ne preclude l’uso come vaccino attenuato.
Avendo stabilito che il picco SHC014 ha la capacità di mediare l’infezione delle cellule umane e causare la malattia nei topi, abbiamo successivamente sintetizzato un clone infettivo SHC014-CoV a lunghezza intera basato sull’approccio utilizzato per SARS-CoV ( Fig. 3a ) 2 . La replica nelle cellule Vero non ha rivelato deficit per SHC014-CoV rispetto a quello per SARS-CoV ( Fig. 3b); tuttavia, SHC014-CoV è stato significativamente attenuato ( P Fig. 3c). L’ infezione in vivo dei topi non ha dimostrato una significativa perdita di peso ma ha mostrato una replicazione virale ridotta nei polmoni dell’infezione SHC014-CoV a lunghezza intera, rispetto alla SARS-CoV Urbani ( Fig. 3d, e). Insieme, i risultati stabiliscono la vitalità del SHC014-CoV a tutta lunghezza, ma suggeriscono che è necessario un ulteriore adattamento affinché la sua replicazione sia equivalente a quella del SARS-CoV epidemico nelle cellule respiratorie umane e nelle cavie ( topi).
Figura 3: SHC014-CoV a lunghezza intera si replica nelle vie aeree umane ma manca della virulenza dell’epidemia di SARS-CoV.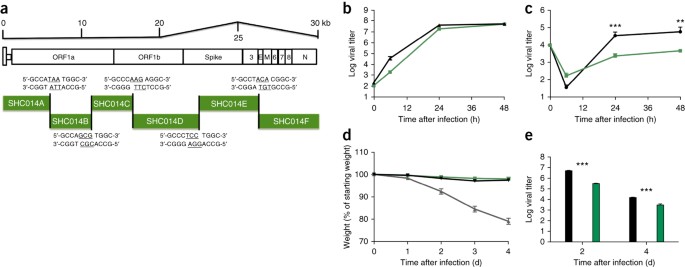
( a ) Schema del clone molecolare SHC014-CoV, che è stato sintetizzato come sei cDNA contigui (designati SHC014A, SHC014B, SHC014C, SHC014D, SHC014E e SHC014F) affiancati da siti BglI unici che consentivano l’assemblaggio diretto dell’espressione cDNA a lunghezza intera frame di lettura aperti (per 1a, 1b, picco, 3, inviluppo, matrice, 6–8 e nucleocapside). I nucleotidi sottolineati rappresentano le sequenze di sporgenza formate dopo la scissione degli enzimi di restrizione. ( b , c ) Replicazione virale di SARS-CoV Urbani (nero) o SHC014-CoV (verde) dopo infezione di cellule Vero ( b ) o colture cellulari HAE con interfaccia aria-liquido primarie ben differenziate ( c) con un MOI di 0,01. I campioni sono stati raccolti in singoli punti temporali con repliche biologiche ( n = 3) per ciascun gruppo. I dati rappresentano un esperimento per entrambe le celle Vero e HAE. ( d , e ) Perdita di peso ( n = 3 per SARS-CoV MA15, n = 7 per SHC014-CoV; n = 6 per SARS-Urbani) ( d ) e replicazione virale nei polmoni ( n = 3 per SARS-Urbani e SHC014-CoV) ( e ) di cavie BALB / c di 10 settimane infettati con 1 × 10 5 pfu di SARS-CoV MA15 (grigio), SHC014-CoV (verde) o SARS-CoV Urbani (nero) tramite in rotta. Ogni punto dati rappresenta la media del gruppo e le barre di errore definiscono il sem **P
Durante l’epidemia di SARS-CoV, furono rapidamente stabiliti collegamenti tra zibetti di palma e ceppi di CoV rilevati nell’uomo 4 . Basandosi su questa scoperta, il paradigma di emergenza comune sostiene che l’epidemia di SARS-CoV ha avuto origine come virus di pipistrello, è passata agli zibetti e ha incorporato i cambiamenti all’interno del dominio di legame dei recettori (RBD) per migliorare il legame con lo zibetto Ace2 (rif. 18 ). La successiva esposizione alle persone nei mercati di animali vivi ha permesso l’infezione umana con il ceppo di zibetto, che, a sua volta, si è adattato per diventare il ceppo epidemico ( Fig. 4a ). Tuttavia, l’analisi filogenetica suggerisce che i primi ceppi di SARS umana appaiono più strettamente correlati ai ceppi di pipistrello che a quelli di zibetto 18. Pertanto, un secondo paradigma sostiene che la trasmissione diretta pipistrello-umano ha avviato l’emergenza SARS-CoV e che gli zibetti di palma sono serviti da ospite secondario e serbatoio per l’infezione continua ( Fig. 4b ) 19 . Per entrambi i paradigmi, l’adattamento del picco in un ospite secondario è visto come una necessità, con la maggior parte delle mutazioni che dovrebbero verificarsi all’interno dell’RBD, facilitando così una migliore infezione. Entrambe le teorie implicano che i pool di CoV di pipistrelli sono limitati e che le mutazioni del raggio ospite sono sia casuali che rare, riducendo la probabilità di futuri eventi di emergenza negli esseri umani.
Figura 4: paradigmi di emergenza per coronavirus.
I ceppi di coronavirus sono mantenuti in pool di quasi specie circolanti nelle popolazioni di pipistrelli. ( a , b ) Le teorie sull’emergenza SARS-CoV tradizionale affermano che i mutanti del raggio ospite (cerchio rosso) rappresentano eventi casuali e rari che consentono l’infezione di ospiti alternativi. Il paradigma dell’ospite secondario ( a ) sostiene che un ospite non umano è infetto da un virus progenitore del pipistrello e, attraverso l’adattamento, facilita la trasmissione all’uomo; la successiva replicazione nell’uomo porta al ceppo virale epidemico. Il paradigma diretto ( b) suggerisce che la trasmissione avviene tra pipistrelli e umani senza la necessità di un ospite intermedio; la selezione si verifica quindi nella popolazione umana con virus strettamente correlati che si replicano in un ospite secondario, consentendo la persistenza e l’adattamento virali continui in entrambi. ( c ) I dati dei virus chimerici simili alla SARS sostengono che i pool di quasi-specie mantengono più virus in grado di infettare le cellule umane senza la necessità di mutazioni (cerchi rossi). Sebbene gli adattamenti negli ospiti secondari o umani possano essere necessari per l’emergenza epidemica, se i virus contenenti picchi SHC014 ricombinati con backbone virulenti di CoV (cerchi con contorni verdi), la malattia epidemica può essere il risultato nell’uomo. Elementi di supporto dati esistenti di tutti e tre i paradigmi.
Sebbene il nostro studio non invalidi le altre vie di emergenza, sostiene un terzo paradigma in cui i pool CoV di pipistrelli circolanti mantengono proteine di picchi “in bilico” in grado di infettare l’uomo senza mutazione o adattamento ( Fig. 4c). Questa ipotesi è illustrata dalla capacità di un virus chimerico contenente il picco SHC014 in una spina dorsale SARS-CoV di causare una infezione robusta in entrambe le colture delle vie aeree umane e nei topi senza adattamento RBD.
Insieme all’osservazione di backbone 3 , 20 di CoV patogeni precedentemente identificati , i nostri risultati suggeriscono che i materiali di partenza richiesti per ceppi emergenti simili alla SARS circolano attualmente in riserve di animali. In particolare, sebbene SHC014-CoV a tutta lunghezza probabilmente richieda un ulteriore adattamento della spina dorsale per mediare la malattia umana, gli eventi di ricombinazione ad alta frequenza documentati nelle famiglie CoV sottolineano la possibilità di emergenze future e la necessità di ulteriore preparazione.
Ad oggi, gli schermi di genomica delle popolazioni animali sono stati utilizzati principalmente per identificare nuovi virus nelle impostazioni dell’epidemia 21 . L’approccio qui estende questi set di dati per esaminare le questioni relative all’emergenza virale e all’efficacia terapeutica. Consideriamo i virus con il picco SHC014 una potenziale minaccia a causa della loro capacità di replicarsi nelle colture primarie delle vie aeree umane, il miglior modello disponibile per le malattie umane. Inoltre, la patogenesi osservata nei topi indica una capacità dei virus contenenti SHC014 di causare malattie nei modelli di mammiferi, senza adattamento RBD.
In particolare, il tropismo differenziale nel polmone rispetto a quello con SARS-MA15 e l’attenuazione di SHC014-CoV a lunghezza intera nelle colture HAE rispetto a SARS-CoV Urbani suggeriscono che fattori oltre il legame ACE2, tra cui la processività dei picchi, la biodisponibilità dei recettori o l’antagonismo delle risposte immunitarie dell’ospite – può contribuire all’emergenza. Tuttavia, sono necessari ulteriori test sui primati non umani per tradurre questi risultati nel potenziale patogeno nell’uomo. È importante sottolineare che il fallimento delle terapie disponibili definisce una necessità fondamentale per ulteriori studi e per lo sviluppo di trattamenti. Con questa conoscenza, possono essere prodotti programmi di sorveglianza, reagenti diagnostici e trattamenti efficaci che sono protettivi contro l’emergenza di CoV specifici del gruppo 2b, come SHC014,
Oltre a offrire preparazione contro i futuri virus emergenti, questo approccio deve essere preso in considerazione nel contesto della pausa del governo degli Stati Uniti sugli studi sul guadagno di funzione (GOF) 22 . Sulla base dei precedenti modelli di emergenza ( Fig. 4a, b ), la creazione di virus chimerici come SHC014-MA15 non avrebbe dovuto aumentare la patogenicità. Sebbene SHC014-MA15 sia attenuato rispetto al suo SARS-CoV adattato dal topo parentale, studi simili che esaminano la patogenicità dei CoV con il picco di tipo Urbani selvatico all’interno della spina dorsale MA15 non hanno mostrato perdita di peso nei topi e ridotta replicazione virale 23 . Pertanto, rispetto al picco Urbani – MA15 CoV, SHC014-MA15 mostra un guadagno nella patogenesi ( Fig. 1). Sulla base di questi risultati, i gruppi di esperti scientifici possono ritenere che studi simili costruiscano virus chimerici basati su ceppi circolanti troppo rischiosi da perseguire, dal momento che non è possibile escludere un aumento della patogenicità nei modelli di mammiferi.
Insieme alle restrizioni sui ceppi adattati al topo e allo sviluppo di anticorpi monoclonali che usano mutanti di fuga, la ricerca sull’emergenza di CoV e l’efficacia terapeutica possono essere fortemente limitate in avanti. Insieme, questi dati e restrizioni rappresentano un crocevia di preoccupazioni per la ricerca del GOF; il potenziale per prepararsi e mitigare i futuri focolai deve essere valutato rispetto al rischio di creare agenti patogeni più pericolosi. Nello sviluppo di politiche che vanno avanti, è importante considerare il valore dei dati generati da questi studi e se questi tipi di studi sui virus chimerici giustificano ulteriori indagini rispetto ai rischi inerenti.
Nel complesso, il nostro approccio ha utilizzato i dati della metagenomica per identificare una potenziale minaccia rappresentata dal CoV SHC014 simile a SARS a mazza circolante. A causa della capacità dei virus chimerici SHC014 di replicarsi nelle culture delle vie respiratorie umane, causare patogenesi in vivo e sfuggire alle attuali terapie, sono necessarie sia la sorveglianza che una migliore terapia contro i virus circolanti simili alla SARS. Il nostro approccio sblocca anche l’uso dei dati metagenomici per prevedere l’emergenza virale e applicare queste conoscenze nella preparazione per il trattamento delle future infezioni da virus emergenti.
METODI
Virus, cellule, infezione in vitro e test sulla placca.
SARS-CoV di tipo selvaggio (Urbani), SARS-CoV adattato per cavie (MA15) e CoV simili a SARS chimerici sono stati coltivati su cellule Vero E6 (ottenute dall’Istituto di ricerca medica dell’esercito degli Stati Uniti), coltivate nell’aquila modificata di Dulbecco medium (DMEM) (Gibco, CA) e 5% siero di clone fetale (FCS) (ciclone, South Logan, UT) insieme ad antibiotico / antimicotico (Gibco, Carlsbad, CA). Le cellule DBT (laboratorio barico, fonte sconosciuta) che esprimono ortologi ACE2 sono state precedentemente descritte sia per l’uomo che per lo zibetto; la sequenza del pipistrello Ace2 si basava su quella del Rhinolophus leschenaulti e le cellule DBT che esprimevano il pipistrello Ace2 erano state stabilite come precedentemente descritto 8 .
Gli esperimenti di pseudotipo erano simili a quelli che utilizzavano uno pseudovirus a base di HIV, preparati come precedentemente descritto 10 ed esaminati su cellule HeLa (Wuhan Institute of Virology) che esprimevano gli ortologi ACE2 . Le cellule HeLa sono state coltivate in mezzo essenziale minimo (MEM) (Gibco, CA) integrato con FCS al 10% (Gibco, CA) come precedentemente descritto 24 .
Le curve di crescita in Vero E6, DBT, Calu-3 2B4 e cellule epiteliali primarie delle vie aeree umane sono state eseguite come precedentemente descritto 8 , 25 . Nessuno degli stock di linee cellulari funzionanti è stato recentemente autenticato o testato per micoplasma, sebbene gli stock di semi originali utilizzati per creare gli stock di lavoro siano privi di contaminazione. I polmoni umani per le colture di HAE sono stati acquistati secondo i protocolli approvati dall’Università del North Carolina presso il Chapel Hill Institutional Review Board. Le colture HAE rappresentano epitelio delle vie aeree umane altamente differenziato contenente cellule epiteliali ciliate e non ciliate e cellule caliciformi. Le colture vengono anche coltivate su un’interfaccia aria-liquido per diverse settimane prima dell’uso, come precedentemente descritto 26 .
In breve, le cellule sono state lavate con PBS e inoculate con virus o diluite in PBS per 40 minuti a 37 ° C. Dopo l’inoculazione, le cellule sono state lavate tre volte e il mezzo fresco è stato aggiunto per indicare il tempo ‘0’. Tre o più replicati biologici sono stati raccolti in ciascun punto temporale descritto. Nessun accecamento è stato usato in nessuna raccolta di campioni né i campioni sono stati randomizzati. Tutta la coltivazione del virus è stata eseguita in un laboratorio di livello di biosicurezza (BSL) 3 con ventilatori ridondanti negli armadi di biosicurezza, come descritto in precedenza dal nostro gruppo 2 . Tutto il personale indossava respiratori a purificazione d’aria potenziati (Breathe Easy, 3M) con tute, grembiuli e stivaletti in Tyvek a doppio guanto.
Clustering di sequenze e modellazione strutturale.
Le sequenze genomiche a lunghezza intera e le sequenze di amminoacidi dei domini S1 del picco di CoV rappresentativi sono state scaricate da Genbank o Pathosystems Resource Integration Center (PATRIC), allineate a ClustalX e filogeneticamente confrontate utilizzando la stima della massima verosimiglianza usando 100 bootstraps o da utilizzando il pacchetto PhyML ( https://code.google.com/p/phyml/ ), rispettivamente. L’albero è stato generato utilizzando la massima probabilità con il pacchetto PhyML. La barra della scala rappresenta le sostituzioni nucleotidiche. Solo i nodi con supporto bootstrap superiore al 70% sono etichettati.
L’albero mostra che i CoV sono divisi in tre distinti gruppi filogenetici definiti come α-CoV, β-CoV e γ-CoV. I cluster di sottogruppi classici sono contrassegnati come 2a, 2b, 2c e 2d per i β-CoV e 1a e 1b per i α-CoV. I modelli strutturali sono stati generati utilizzando Modeller (Max Planck Institute Bioinformatics Toolkit) per generare modelli di omologia per SHC014 e Rs3367 del SARS RBD in complesso con ACE2 basato sulla struttura cristallina 2AJF (Protein Data Bank). I modelli di omologia sono stati visualizzati e manipolati in MacPyMol (versione 1.3).
Costruzione di virus chimerici simili alla SARS.
Sia i virus wild-type che i virus chimerici sono derivati da SARS-CoV Urbani o dal corrispondente clone infettivo (icS) adattato alla cavia (SARS-CoV MA15) come precedentemente descritto 27 . I plasmidi contenenti sequenze di picchi per SHC014 sono stati estratti mediante digestione di restrizione e legati nel plasmide E ed F del clone infettivo MA15. Il clone è stato progettato e acquistato da Bio Basic come sei cDNA contigui utilizzando sequenze pubblicate affiancate da siti unici di endonucleasi di restrizione di classe II (BglI). Successivamente, i plasmidi contenenti frammenti di genoma SARS-CoV di tipo selvaggio, chimerico e SHC014-CoV sono stati amplificati, escisi, ligati e purificati.
Le reazioni di trascrizione in vitro sono state quindi preformate per sintetizzare l’RNA genomico a lunghezza intera, che è stato trasfettato nelle cellule Vero E6 come precedentemente descritto 2 . Il mezzo proveniente da cellule trasfettate è stato raccolto e servito come stock di semi per successivi esperimenti. I virus chimerici e full-length sono stati confermati dall’analisi della sequenza prima dell’uso in questi studi. La costruzione sintetica di mutante chimerico e SHC014-CoV integrale è stata approvata dal Comitato istituzionale per la biosicurezza dell’Università del Nord Carolina e dal Comitato per la Ricerca del Doppio Uso.
Dichiarazione etica.
Questo studio è stato condotto in conformità con le raccomandazioni per la cura e l’uso degli animali da parte dell’Office of Laboratory Animal Welfare (OLAW), NIH. Il Comitato Istituzionale per la Cura e l’Uso degli Animali (IACUC) dell’Università della Carolina del Nord a Chapel Hill (UNC, permesso numero A-3410-01) ha approvato il protocollo di studio sugli animali (IACUC n. 13-033) utilizzato in questi studi.
Cavie e infezione in vivo .
Topi femmina, 10 settimane e 12 mesi BALB / cAnNHsD sono stati ordinati da Harlan Laboratories. Le infezioni da topo sono state fatte come precedentemente descritto 20 . In breve, gli animali sono stati portati in un laboratorio BSL3 e lasciato acclimatarsi per 1 settimana prima dell’infezione. Per l’infezione e la vaccinazione virale attenuata dal vivo, i topi sono stati anestetizzati con una miscela di ketamina e xilazina e infettati per via intranasale, quando sottoposti a contestazione, con 50 μl di soluzione salina tamponata con fosfato (PBS) o virus diluito con tre o quattro topi per volta, per gruppo di infezione per dose come descritto nella figura legende.
Per i singoli topi, le notazioni di infezione tra cui la mancata inalazione dell’intera dose, il gorgoglio dell’inoculo dal naso o l’infezione attraverso la bocca possono aver portato all’esclusione dei dati del topo a discrezione del ricercatore; post-infezione, non sono definiti altri criteri di esclusione o inclusione prestabiliti. Nessun accecamento è stato usato in nessun esperimento su animali e gli animali non sono stati randomizzati. Per la vaccinazione, topi giovani e anziani sono stati vaccinati mediante iniezione della zampa con un volume di 20 μl di 0,2 μg di vaccino SARS-CoV a doppia inattivazione con allume o PBS finto; i topi sono stati quindi potenziati con lo stesso regime 22 giorni dopo e sfidati 21 giorni dopo. Per tutti i gruppi, come da protocollo, gli animali sono stati monitorati quotidianamente per rilevare segni clinici di malattia (ingobbimento, pelo arruffato e attività ridotta) per la durata dell’esperimento.
Tutti i topi che hanno perso oltre il 20% del loro peso corporeo iniziale sono stati nutriti a terra e monitorati ulteriormente più volte al giorno, purché fossero sotto il limite del 20%. I topi che hanno perso oltre il 30% del loro peso corporeo iniziale sono stati immediatamente sacrificati secondo il protocollo. Qualsiasi topo ritenuto moribondo o improbabile da recuperare è stato anche umanamente sacrificato a discrezione del ricercatore. L’eutanasia è stata eseguita utilizzando un sovradosaggio di isoflurano e la morte è stata confermata dalla lussazione cervicale. Tutti gli studi sui topi sono stati condotti presso l’Università della Carolina del Nord (Animal Welfare Assurance n. A3410-01) utilizzando protocolli approvati dal Comitato istituzionale per la cura e l’uso degli animali dell’UNC (IACUC).
Analisi istologica
Il polmone sinistro è stato rimosso e immerso in formalina tamponata al 10% (Fisher) senza inflazione per 1 settimana. I tessuti sono stati incorporati nella paraffina e sezioni di 5 micron sono state preparate dalla struttura di base istopatologia del Centro per il Cancro Globale Lineberger dell’UNC. Per determinare l’estensione della colorazione dell’antigene, le sezioni sono state colorate per l’antigene virale usando un anticorpo anti-nucleocapside SARS-CoV policlonale disponibile in commercio (Imgenex) e marcato in modo cieco per la colorazione delle vie aeree e del parenchima come precedentemente descritto 20 . Le immagini sono state catturate utilizzando un microscopio Olympus BX41 con una fotocamera Olympus DP71.
Saggi di neutralizzazione dei virus.
I saggi del titolo di neutralizzazione della riduzione della placca sono stati eseguiti con anticorpi precedentemente caratterizzati contro SARS-CoV, come precedentemente descritto 11 , 12 , 13 . In breve, gli anticorpi o il siero neutralizzanti sono stati diluiti in serie due volte e incubati con 100 pfu dei diversi ceppi SARS-CoV del clone infettivo per 1 ora a 37 ° C. Il virus e gli anticorpi sono stati quindi aggiunti a una piastra da 6 pozzetti con 5 × 10 5 cellule Vero E6 / pozzetto con repliche multiple ( n ≥ 2). Dopo un’incubazione di 1 ora a 37 ° C, le cellule sono state sovrapposte con 3 ml di agarosio allo 0,8% in media. Le piastre sono state incubate per 2 giorni a 37 ° C, colorate con rosso neutro per 3 ore e sono state contate le placche. La percentuale di riduzione della placca è stata calcolata come (1 – (n. Di placche con anticorpo / n. Di placche senza anticorpo)) × 100.
Analisi statistica.
Tutti gli esperimenti sono stati condotti contrastando due gruppi sperimentali (due virus o coorti vaccinati e non vaccinati). Pertanto, differenze significative nel titolo virale e nel punteggio istologico sono state determinate dal test t di uno Studente a due code nei singoli punti temporali. I dati venivano normalmente distribuiti in ciascun gruppo confrontato e presentavano una varianza simile.
Biosicurezza e bioprotezione.
Sono stati avviati studi segnalati dopo che il Comitato Istituzionale per la Biosicurezza dell’Università del Nord Carolina ha approvato il protocollo sperimentale (Titolo del progetto: Generazione di cloni infetti di COV simili a SAR di pipistrello; ID piano di sicurezza di laboratorio: 20145741; ID G di pianificazione: 12279). Questi studi sono stati avviati prima della pausa del finanziamento della ricerca sui processi deliberativi del governo degli Stati Uniti sulla ricerca di guadagno funzionale selezionata che coinvolge virus influenzali, MERS e SARS ( http://www.phe.gov/s3/dualuse/Documents/gain-of-function .pdf ). Questo documento è stato esaminato dall’agenzia di finanziamento, NIH. È stato richiesto il proseguimento di questi studi, che è stato approvato dal NIH.
SARS-CoV è un agente selezionato. Tutto il lavoro per questi studi è stato eseguito con procedure operative standard approvate (SOP) e condizioni di sicurezza per SARS-CoV, MERs-CoV e altri CoV correlati. Le nostre strutture istituzionali BSL3 CoV sono state progettate per conformarsi ai requisiti di sicurezza raccomandati nei laboratori di biosicurezza nei laboratori microbiologici e biomedici (BMBL), nel Dipartimento della salute e dei servizi umani degli Stati Uniti, nel Servizio sanitario pubblico, nei Centri per il controllo delle malattie (CDC ) e il NIH. Sono stati presentati piani di sicurezza di laboratorio e la struttura è stata approvata per l’uso da parte del Dipartimento di salute e sicurezza ambientale (EHS) dell’UNC e del CDC. L’accesso alla scheda elettronica è necessario per l’ingresso nella struttura.
Tutti i lavoratori sono stati formati da EHS per utilizzare in modo sicuro respiratori alimentati a purificazione dell’aria (PAPR), e sono in atto adeguate abitudini di lavoro in una struttura BSL3 e sono attivi piani di sorveglianza medica attiva. Le nostre strutture CoL BSL3 contengono ventole ridondanti, alimentazione di emergenza per ventilatori e armadi e congelatori biologici di sicurezza, e le nostre strutture possono ospitare rack per mouse SealSafe. I materiali classificati come agenti BSL3 sono costituiti da SARS-CoV, ceppi precursori di bat CoV, MERS-CoV e mutanti derivati da questi agenti patogeni. All’interno delle strutture BSL3, la sperimentazione con un virus infettivo viene eseguita in un armadio di biosicurezza di classe II (BSC) certificato.
Tutti i membri del personale indossano scrub, tute e grembiuli Tyvek, PAPR e copriscarpe e le loro mani sono a doppio guanto. Gli utenti di BSL3 sono soggetti a un piano di sorveglianza medica monitorato dalla Clinica per la Salute sul Lavoro dei Dipendenti dell’Università (UEOHC), che include una vaccinazione antinfluenzale annuale e fisica annuale e la segnalazione obbligatoria di tutti i sintomi associati all’infezione da CoV durante i periodi di lavoro nel BSL3. Tutti gli utenti di BSL3 sono addestrati nella gestione dell’esposizione e nei protocolli di segnalazione, sono preparati all’auto-quarantena e sono stati addestrati per la consegna sicura a un dipartimento locale di gestione delle malattie infettive in una situazione di emergenza. Tutti i potenziali eventi di esposizione sono segnalati e indagati da EHS e UEOHC, con segnalazioni presentate sia al CDC che al NIH.
Accession codes
Accessions
Protein Data Bank
Change history
20 November 2015
In the version of this article initially published online, the authors omitted to acknowledge a funding source, USAID-EPT-PREDICT funding from EcoHealth Alliance, to Z.-L.S. The error has been corrected for the print, PDF and HTML versions of this article.
References
- 1Ge, X.Y. et al. Isolation and characterization of a bat SARS-like coronavirus that uses the ACE2 receptor. Nature 503, 535–538 (2013).
- 2Yount, B. et al. Reverse genetics with a full-length infectious cDNA of severe acute respiratory syndrome coronavirus. Proc. Natl. Acad. Sci. USA 100, 12995–13000 (2003).
- 3Becker, M.M. et al. Synthetic recombinant bat SARS-like coronavirus is infectious in cultured cells and in mice. Proc. Natl. Acad. Sci. USA 105, 19944–19949 (2008).
- 4Peiris, J.S., Guan, Y. & Yuen, K.Y. Severe acute respiratory syndrome. Nat. Med. 10, S88–S97 (2004).
- 5Al-Tawfiq, J.A. et al. Surveillance for emerging respiratory viruses. Lancet Infect. Dis. 14, 992–1000 (2014).
- 6He, B. et al. Identification of diverse alphacoronaviruses and genomic characterization of a novel severe acute respiratory syndrome–like coronavirus from bats in China. J. Virol. 88, 7070–7082 (2014).
- 7Li, F. Receptor recognition and cross-species infections of SARS coronavirus. Antiviral Res. 100, 246–254 (2013).
- 8Sheahan, T. et al. Mechanisms of zoonotic severe acute respiratory syndrome coronavirus host range expansion in human airway epithelium. J. Virol. 82, 2274–2285 (2008).
- 9Yoshikawa, T. et al. Dynamic innate immune responses of human bronchial epithelial cells to severe acute respiratory syndrome–associated coronavirus infection. PLoS ONE 5, e8729 (2010).
- 10Qiu, X. et al. Reversion of advanced Ebola virus disease in nonhuman primates with ZMapp. Nature 514, 47–53 (2014).
- 11Sui, J. et al. Broadening of neutralization activity to directly block a dominant antibody-driven SARS-coronavirus evolution pathway. PLoS Pathog. 4, e1000197 (2008).
- 12Sui, J. et al. Effects of human anti–spike protein receptor binding domain antibodies on severe acute respiratory syndrome coronavirus neutralization escape and fitness. J. Virol. 88, 13769–13780 (2014).
- 13Rockx, B. et al. Escape from human monoclonal antibody neutralization affects in vitro and in vivo fitness of severe acute respiratory syndrome coronavirus. J. Infect. Dis. 201, 946–955 (2010).
- 14Spruth, M. et al. A double-inactivated whole-virus candidate SARS coronavirus vaccine stimulates neutralizing and protective antibody responses. Vaccine 24, 652–661 (2006).
- 15Bolles, M. et al. A double-inactivated severe acute respiratory syndrome coronavirus vaccine provides incomplete protection in mice and induces increased eosinophilic proinflammatory pulmonary response upon challenge. J. Virol. 85, 12201–12215 (2011).
- 16Siegrist, C.-A. in Vaccines 6th edn. (eds. Plotkin, S.A., Orenstein, W.A. & Offit, P.A.) 14–32 (W.B. Saunders, 2013).
- 17Deming, D. et al. Vaccine efficacy in senescent mice challenged with recombinant SARS-CoV bearing epidemic and zoonotic spike variants. PLoS Med. 3, e525 (2006).
- 18Graham, R.L., Donaldson, E.F. & Baric, R.S. A decade after SARS: strategies for controlling emerging coronaviruses. Nat. Rev. Microbiol. 11, 836–848 (2013).
- 19Graham, R.L. & Baric, R.S. Recombination, reservoirs and the modular spike: mechanisms of coronavirus cross-species transmission. J. Virol. 84, 3134–3146 (2010).
- 20Agnihothram, S. et al. A mouse model for betacoronavirus subgroup 2c using a bat coronavirus strain HKU5 variant. MBio 5, e00047-14 (2014).
- 21Relman, D.A. Metagenomics, infectious disease diagnostics and outbreak investigations: sequence first, ask questions later? J. Am. Med. Assoc. 309, 1531–1532 (2013).
- 22Kaiser, J. Moratorium on risky virology studies leaves work at 14 institutions in limbo. ScienceInsider http://news.sciencemag.org/biology/2014/11/moratorium-risky-virology-studies-leaves-work-14-institutions-limbo (2014).
- 23Frieman, M. et al. Molecular determinants of severe acute respiratory syndrome coronavirus pathogenesis and virulence in young and aged mouse models of human disease. J. Virol. 86, 884–897 (2012).
- 24Ren, W. et al. Difference in receptor usage between severe acute respiratory syndrome (SARS) coronavirus and SARS-like coronavirus of bat origin. J. Virol. 82, 1899–1907 (2008).
- 25Sims, A.C. et al. Release of severe acute respiratory syndrome coronavirus nuclear import block enhances host transcription in human lung cells. J. Virol. 87, 3885–3902 (2013).
- 26Fulcher, M.L., Gabriel, S., Burns, K.A., Yankaskas, J.R. & Randell, S.H. Well-differentiated human airway epithelial cell cultures. Methods Mol. Med. 107, 183–206 (2005).
- 27Roberts, A. et al. A mouse-adapted SARS-coronavirus causes disease and mortality in BALB/c mice. PLoS Pathog. 3, e5.
Acknowledgements
Research in this manuscript was supported by grants from the National Institute of Allergy & Infectious Disease and the National Institute of Aging of the US National Institutes of Health (NIH) under awards U19AI109761 (R.S.B.), U19AI107810 (R.S.B.), AI085524 (W.A.M.), F32AI102561 (V.D.M.) and K99AG049092 (V.D.M.), and by the National Natural Science Foundation of China awards 81290341 (Z.-L.S.) and 31470260 (X.-Y.G.), and by USAID-EPT-PREDICT funding from EcoHealth Alliance (Z.-L.S.). Human airway epithelial cultures were supported by the National Institute of Diabetes and Digestive and Kidney Disease of the NIH under award NIH DK065988 (S.H.R.). We also thank M.T. Ferris (Dept. of Genetics, University of North Carolina) for the reviewing of statistical approaches and C.T. Tseng (Dept. of Microbiology and Immunology, University of Texas Medical Branch) for providing Calu-3 cells. Experiments with the full-length and chimeric SHC014 recombinant viruses were initiated and performed before the GOF research funding pause and have since been reviewed and approved for continued study by the NIH. The content is solely the responsibility of the authors and does not necessarily represent the official views of the NIH.
Author information
Affiliations
- Department of Epidemiology, the University of North Carolina at Chapel Hill, Chapel Hill, North Carolina, USA
- Vineet D Menachery
- , Boyd L Yount Jr
- , Kari Debbink
- , Lisa E Gralinski
- , Jessica A Plante
- , Rachel L Graham
- , Trevor Scobey
- , Eric F Donaldson
- & Ralph S Baric
- Department of Microbiology and Immunology, University of North Carolina at Chapel Hill, Chapel Hill, North Carolina, USA
- Kari Debbink
- & Ralph S Baric
- National Center for Toxicological Research, Food and Drug Administration, Jefferson, Arkansas, USA
- Sudhakar Agnihothram
- Key Laboratory of Special Pathogens and Biosafety, Wuhan Institute of Virology, Chinese Academy of Sciences, Wuhan, China
- Xing-Yi Ge
- & Zhengli-Li Shi
- Department of Cell Biology and Physiology, University of North Carolina at Chapel Hill, Chapel Hill, North Carolina, USA
- Scott H Randell
- Cystic Fibrosis Center, Marsico Lung Institute, University of North Carolina at Chapel Hill, Chapel Hill, North Carolina, USA
- Scott H Randell
- Institute for Research in Biomedicine, Bellinzona Institute of Microbiology, Zurich, Switzerland
- Antonio Lanzavecchia
- Department of Cancer Immunology and AIDS, Dana-Farber Cancer Institute, Harvard Medical School, Boston, Massachusetts, USA
- Wayne A Marasco
- Department of Medicine, Harvard Medical School, Boston, Massachusetts, USA
- Wayne A Marasco
Contributions
V.D.M. designed, coordinated and performed experiments, completed analysis and wrote the manuscript. B.L.Y. designed the infectious clone and recovered chimeric viruses; S.A. completed neutralization assays; L.E.G. helped perform mouse experiments; T.S. and J.A.P. completed mouse experiments and plaque assays; X.-Y.G. performed pseudotyping experiments; K.D. generated structural figures and predictions; E.F.D. generated phylogenetic analysis; R.L.G. completed RNA analysis; S.H.R. provided primary HAE cultures; A.L. and W.A.M. provided critical monoclonal antibody reagents; and Z.-L.S. provided SHC014 spike sequences and plasmids. R.S.B. designed experiments and wrote the manuscript.
Corresponding authors
Correspondence to Vineet D Menachery or Ralph S Baric.
Ethics declarations
Competing interests
The authors declare no competing financial interests.
Supplementary Text and Figures
Supplementary Figures 1–6 and Supplementary Tables 1–4 (PDF 4747 kb)Reprints and PermissionsCite this articleMenachery, V., Yount, B., Debbink, K. et al. A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence. Nat Med21, 1508–1513 (2015). https://doi.org/10.1038/nm.3985Download citation
- Received12 June 2015
- Accepted08 October 2015
- Published09 November 2015
- Issue DateDecember 2015
*****